【论文分享】从多模态电子健康记录中学习模态间对应和表型
从多模态电子健康记录中学习模态间对应和表型
| Title | Learning Inter-Modal Correspondence and Phenotypes from Multi-Modal Electronic Health Records |
|---|---|
| Author | Kejing Yin; William K. Cheung; Benjamin C. M. Fung; Jonathan Poon |
| Affiliations | Department of Computer Science, Hong Kong Baptist University; School of Information Studies, McGill University, Montreal, Canada;Hong Kong Hospital Authority, Hong Kong |
| Emails | {cskjyin, william}@comp.hkbu.edu.hk; ben.fung@mcgill.ca; jonathan@ha.org.hk |
| Paper | https://ieeexplore.ieee.org/abstract/document/9261129 |
| Code | https://github. com/jakeykj/cHITF |
摘要
Non-negative tensor factorization has been shown a practical solution to automatically discover phenotypes from the electronic health records (EHR) with minimal human supervision. Such methods generally require an input tensor describing the inter-modal interactions to be pre-established; however, the correspondence between different modalities (e.g., correspondence between medications and diagnoses) can often be missing in practice. Although heuristic methods can be applied to estimate them, they inevitably introduce errors, and leads to sub-optimal phenotype quality. This is particularly important for patients with complex health conditions (e.g., in critical care) as multiple diagnoses and medications are simultaneously present in the records. To alleviate this problem and discover phenotypes from EHR with unobserved inter-modal correspondence, we propose the collective hidden interaction tensor factorization (cHITF) to infer the correspondence between multiple modalities jointly with the phenotype discovery. We assume that the observed matrix for each modality is marginalization of the unobserved inter-modal correspondence, which are reconstructed by maximizing the likelihood of the observed matrices. Extensive experiments conducted on the real-world MIMIC-III dataset demonstrate that cHITF effectively infers clinically meaningful inter-modal correspondence, discovers phenotypes that are more clinically relevant and diverse, and achieves better predictive performance compared with a number of state-of-the-art computational phenotyping models.
非负张量分解已被证明是一种实用的解决方案,可以在尽可能少的人力监督下从电子健康记录( EHR )中自动发现表型。这类方法一般需要预先建立描述模态间相互作用的输入张量;然而,在实际应用中,不同模态(例如,药物与诊断之间的对应关系)之间的对应关系往往会缺失。虽然启发式方法可以用来估计它们,但它们不可避免地引入误差,并导致次优的表型质量。这对于具有复杂健康状况的患者(例如,在重症监护中)尤为重要,因为记录中同时存在多个诊断和药物。为了缓解这一问题,并从未观察到模态间对应关系的EHR中发现表型,我们提出了集体隐藏交互张量分解( cHITF ),与表型发现一起推断多个模态之间的对应关系。我们假设每个模态的观测矩阵是未观测到的模态间对应的边缘化,通过最大化观测矩阵的似然来重建。在真实的MIMIC - III数据集上进行的大量实验表明,与一些最先进的计算表型模型相比,cHITF有效地推断了具有临床意义的模态间对应关系,发现了更具有临床相关性和多样性的表型,并取得了更好的预测性能。
**此篇论文是上篇文章“Joint Learning of Phenotypes and Diagnosis-Medication Correspondence via Hidden Interaction Tensor Factorization”的期刊扩展,在HITF的基础上添加了多个模态进行表型提取。**详情请见:https://mp.weixin.qq.com/s/DOHoCVKl1QacT020GxqWkA。
有关张量分解的表型提取
表型被正式定义为一组高度相关且能更好地表征患者健康状况的临床特征,而表型提取就是从电子健康记录(EHR)中提取出一组表征患者健康状况的临床特征。传统上,表型分析是以监督的方式进行的,涉及到手动标记病例和对照患者,并总结和提炼针对特定疾病的判别特征的迭代过程,这显然是费时费力的。为了加快表型分析的速度,可以应用机器学习方法(特别是无监督的方法),从大规模的EHR数据中自动提取表型。
非负张量CP分解由于其高度的可解释性和保持高阶相互作用的能力应用于表型提取中。下图展示了一个使用CP分解的表型过程的例子。模型的输入是由三个模态组成的张量:病人,诊断和药物。输出是秩一张量的加权和。每个秩一张量由三个因子向量的外积组成,这三个因子向量是通过求解三个模态的优化问题得到的。这些因子向量可以通过模式组织成因子矩阵(权重λ已被吸收到病人因子矩阵中)。
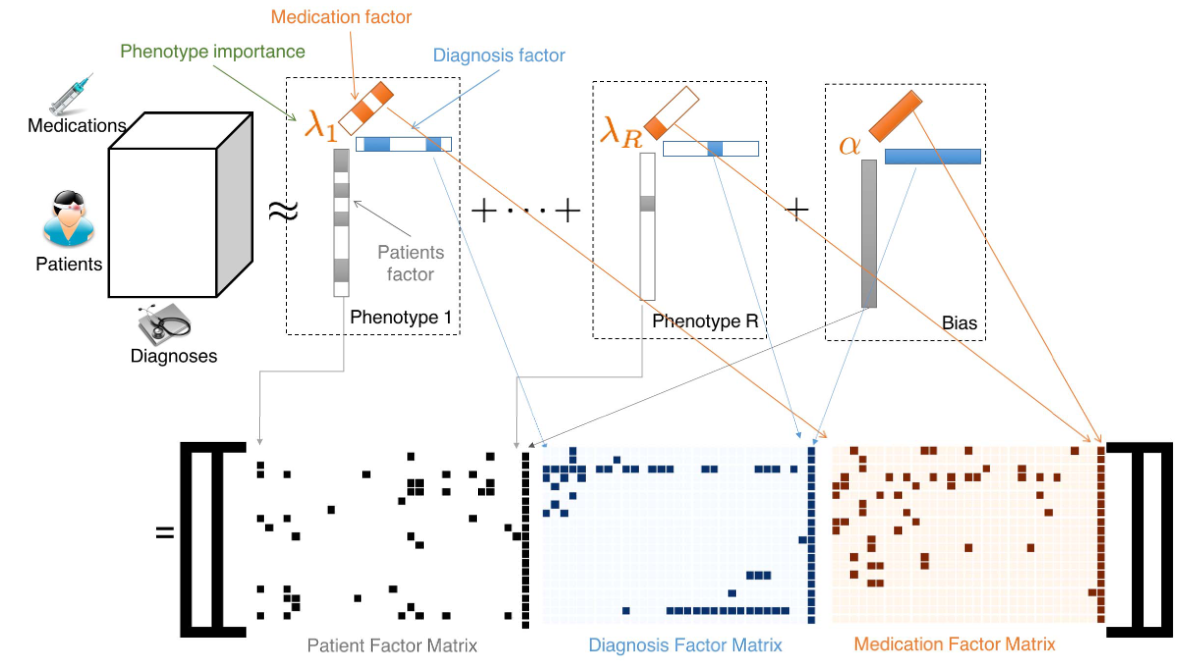
CP-factorization-medication
用数学公式可表示为:
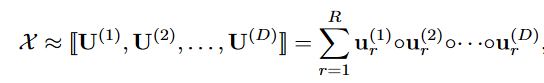
CP-factorization-math
其中U(1),…U(n)是不同模态的因子矩阵。
尽管非负CP分解在表型分析中取得了巨大成功,但仍有一些基本挑战阻碍了它在一些实际场景中的应用,包括:
挑战一:隐性互动(Hidden interactions)。作为包括非负CP分解在内的任何普通张量分解模型的输入,张量需要被很好地定义以表示不同模态之间的相互作用。然而,这些信息在实践中往往无法获得。以诊断和用药为例,EHR数据通常只包含患者的诊断列表和药物处方列表,而药物与诊断之间的对应关系完全没有记录。现有的方法转向一种替代策略,即考虑"共现"关系,即隐含假设在同一临床访问中同时出现的所有药物和诊断将相互对应。这种"等对应"的假设对于某些特定类型的数据集是合理的,例如初级保健或门诊数据,在这些数据集中,患者通常在每次临床访问中都有非常不同的诊断。然而,现实世界中的EHR数据往往是高度复杂的,例如住院或重症监护数据,其中患者通常具有非常复杂的医疗条件:患者可以分配超过十几个诊断代码和数十种药物。在这种情况下,“对等"假设不再成立。
挑战二:多模态(Multi-modality)。EHR前所未有的丰富性通过其多模态性得以展现。除了诊断和药物外,一个典型的EHR数据集( e.g. MIMIC -Ⅲ)可以涉及其他模态,包括程序、实验室检测结果和输入液体。它们各自包含了关于患者不同方面的独特信息,但它们通常具有很强的相关性,并且经常以复杂的方式相互作用,这给在单个张量中建模多模态EHR数据并从中发现表型带来了额外的困难。首先,涉及的模态越多,明确定义模态间的相互作用越困难,需要在此基础上构建输入张量。随着模态数量的增加,张量条目的物理意义变得微妙,可解释性会受到影响。例如,诊断、药物和实验室检查之间的相互作用可能比仅诊断和药物之间的相互作用更加模糊,因为可以要求实验室检查来确认诊断,或者监测药物的使用。此外,张量分解模型的运行时间通常随模态数呈指数增长,这使得其无法扩展到多模态数据。此外,不同的模态往往具有不同的数据类型,例如用于诊断的二进制数据,表明患者记录中存在或不存在诊断代码,而输入到患者的液体量以真实值记录。提高衍生表型质量的关键之一是不同模态的适当组合,以最大限度地提高可解释性。
HITF
在介绍cHITF之前,必须先回顾一下HITF。HITF旨在发现边缘化观测下不同模态项目之间的不可观测对应关系。在普通的CP分解中,因子可以通过最小化重构误差或最大化输入张量的似然来估计。然而,在HITF的设定中,我们只观察到高阶相互作用张量的边缘化。因此,我们通过最大化边缘化的可能性来求解因子,而不是张量X本身。为此,我们首先将同样的边缘化应用于重构,得到沿第n维的边缘化vn如下:
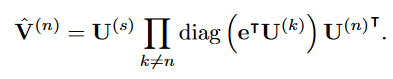
Vn-math
令V表示一组N个观测矩阵,每个观测矩阵对应一个特定的模态。我们假设观测矩阵是通过边缘化描述模态间相互作用的不可观测的高阶张量生成的。作为示例,下图描述了EHR中具有两种模态的HITF模型:用药处方(medication prescriptions)和诊断代码(diagnosis codes)。我们观察两个矩阵:患者-药物(patient-by-medication)矩阵V ( 1 )和患者-诊断(patient-by-diagnosis)矩阵V ( 2 ),分别记录每个患者的处方药物和诊断代码。可以合理地假设这两种模式之间存在某种对应关系——根据某些诊断向患者开出药物。因此,我们假设存在一个具有N + 1个模态的高阶隐相互作用张量X,描述模态间的对应关系,并通过边缘化隐相互作用张量得到观测矩阵V。
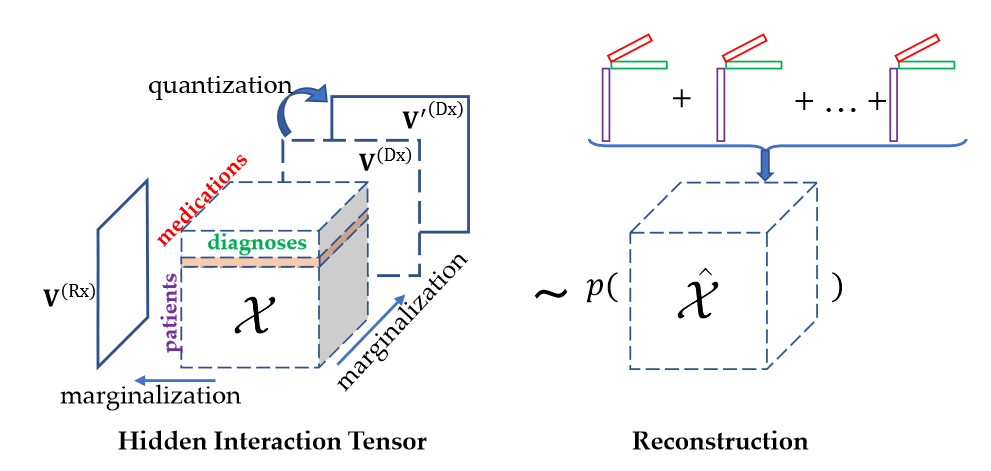
HITF
cHITF
尽管HITF能够对两种以上的模态进行建模,但直接将其应用于多模态EHR数据进行计算表型分析往往会受到如前所述的所有模态之间的对应关系解释困难的阻碍。因此,更适合的方法是发现每两个模态之间的兴趣对应关系,以便更好地解释。例如,给定诊断,药物和实验室检查,我们可以推断诊断与药物、诊断与实验室检查之间分别存在对应关系。为了实现这一点,我们引入集体隐交互张量分解( cHITF )框架来同时学习表型和推断模态之间的对应关系。模型框架图如下图所示。
其中,诊断方式(Dx)在临床数据中起着核心作用,因为药物和液体大多是由诊断决定的;实验室检测结果异常是反映由病理或诊断引起的异常生化状态的指标。显然,它更容易解释诊断与其他三种模态之间的对应关系。因此,我们假设存在三个隐藏的相互作用张量分别捕获诊断和药物之间、诊断和实验室检测之间以及诊断和输入流体之间的对应关系,我们应用HITF来联合求解每一个相互作用的张量。患者模式是所有子问题中的共享维度(对应于U ( Pt ))因子矩阵),由于我们以诊断为中心的设计,诊断模式也是共享的;因此,我们在框架中强制要求它们对应的因子矩阵相同。
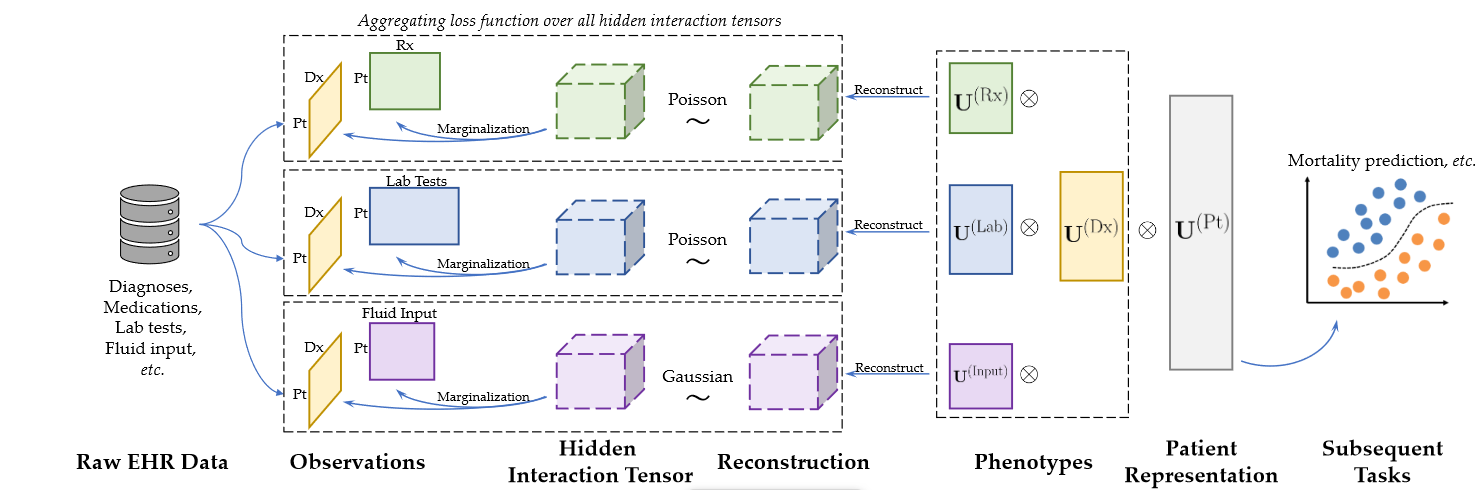
cHITF
模型的建立与求解
损失函数形式表现为如下:
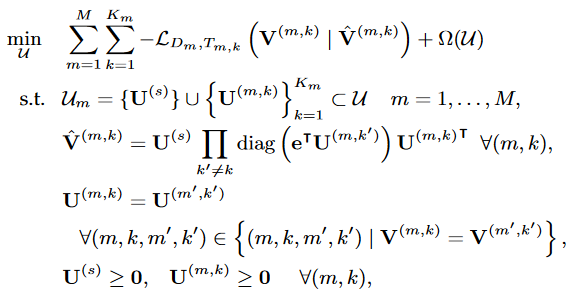
min-math
其中,M是隐藏的相互作用张量的个数,Km为第m个隐交互张量的组成模态数(在上图中,Km=2,m=1,2,3),
该论文尝试了两种数据分布探究表型分解的效果:泊松分布与高斯分布;
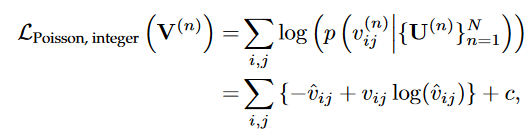
possion
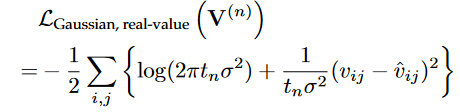
gaussian
该论文通过块坐标下降( BCD )方法优化损失函数。具体来说,在U中的潜因子之间进行交替,并在所有其他潜因子固定的情况下更新每个潜因子,使用投影梯度下降来更新潜在因子。
实验结果
对于提取出的表型是否有意义?
该论文对结果的评估结合了临床专家打分与张量分解获得的表型对应得分,具体表现为:
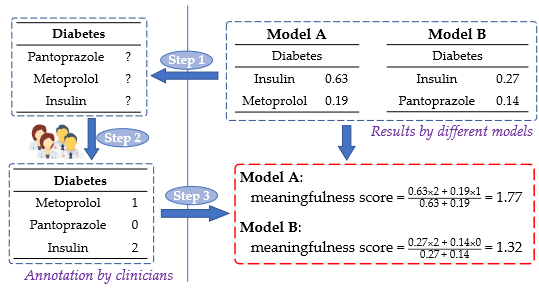
estimate
实验结果:
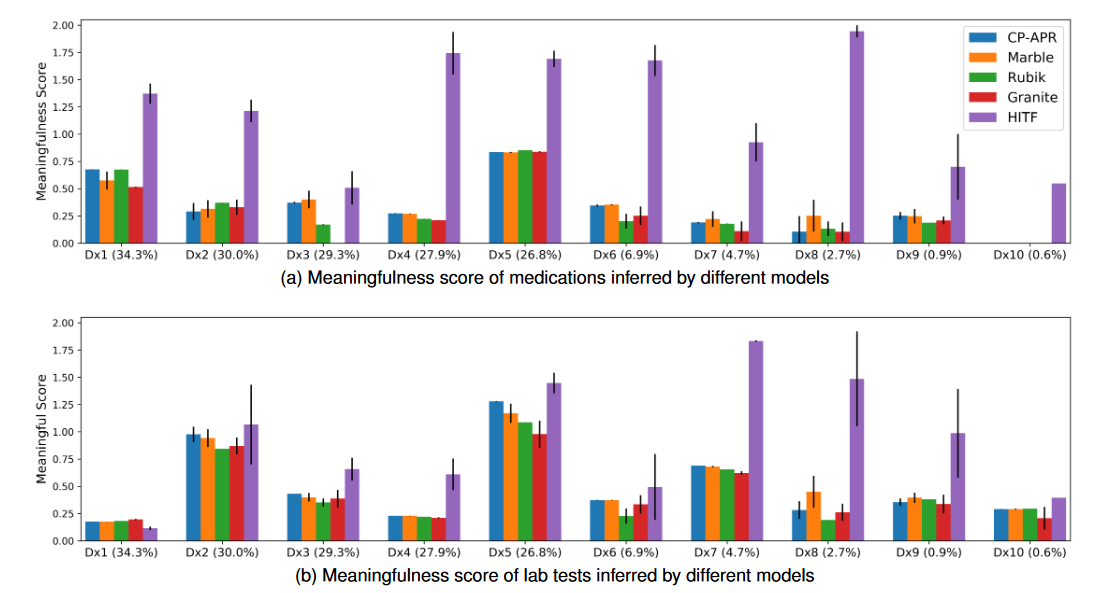
result1
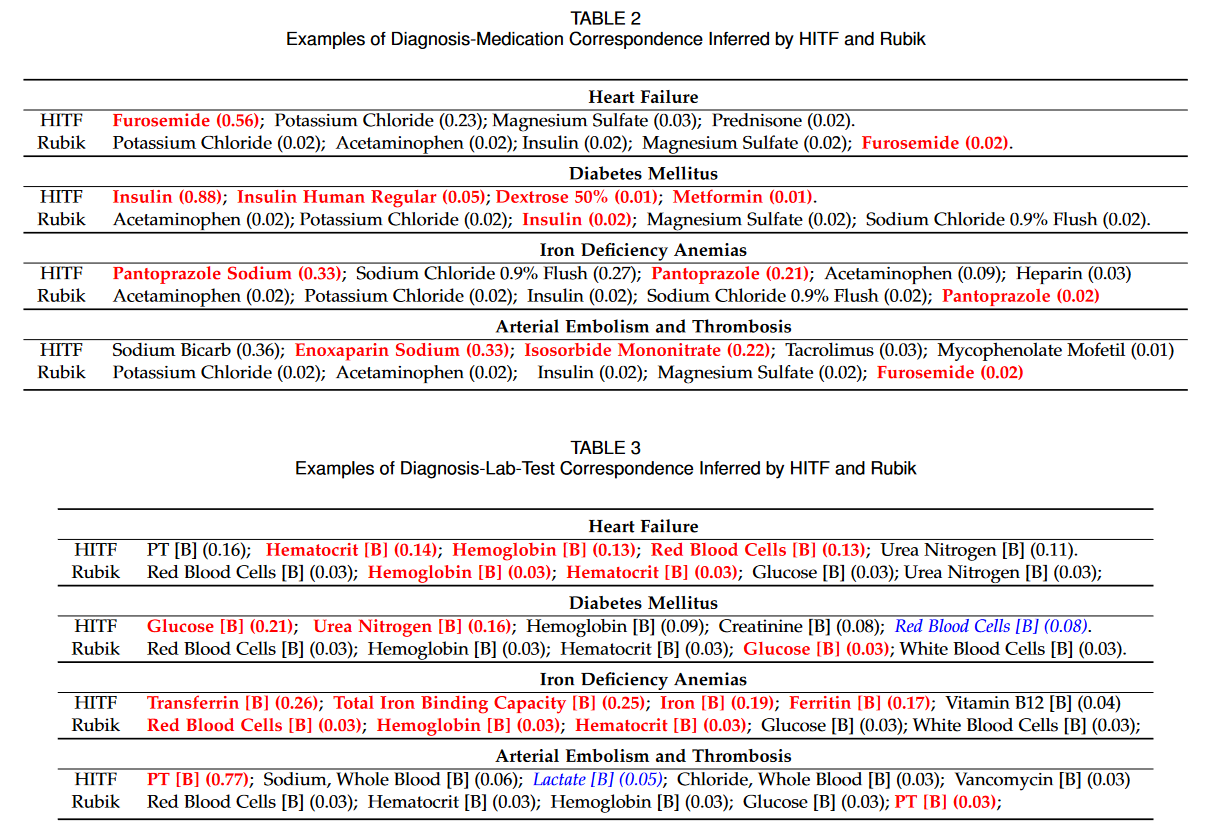
result2
在表2和表3中,每个项目后面的数字是推断的相应分数。由临床医师标注,红色加粗文本中的条目有临床意义,蓝色斜体文本中的条目可能有意义,其余条目无意义。
这篇论文还评估了使用cHITF推断的表型作为特征用于后续住院死亡率预测任务的性能。首先以8:2的比例划分数据集进行训练和测试,并采用五折交叉验证。然后,应用cHITF和所有不同模态的基线来学习训练子集的表型和患者表示。我们将测试子集投影到学习到的表型上,得到测试子集的患者表示。我们使用lasso正则化逻辑回归来执行以患者表示为特征的二分类。我们使用AUPRC (精确率-召回率曲线下面积)作为计算的评价指标。
实验结果:
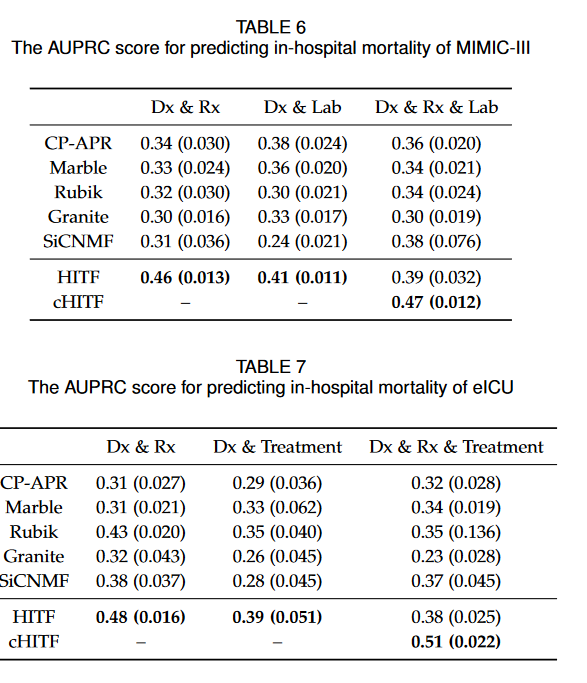
result3
从多模态EHR数据中推断具有临床可解释性和相关性的表型是首要任务,所以该论文还评估了获得的表型的稀疏性和多样性,
多样性的评估指标如下:
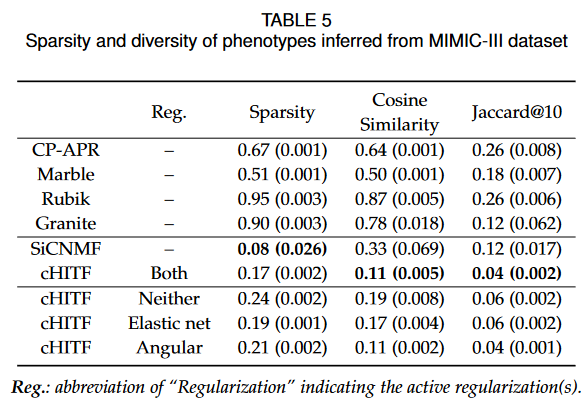
result4
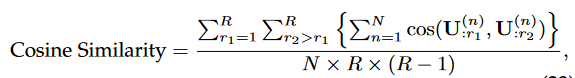
similarity
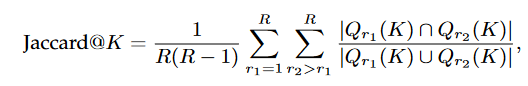
jaccsrd